No supervisado I¶
Clustering¶
Clustering es el proceso de agrupar objetos en conjuntos de acuerdo con su similitud, de modo que los elementos dentro de un mismo grupo compartan características comunes. Es una técnica clave en la minería de datos exploratoria y ampliamente utilizada en el análisis estadístico de datos para descubrir patrones y relaciones ocultas en los datos.

K-means¶
Teoría¶
El algoritmo K-means (MacQueen, 1967) agrupa las observaciones en un número predefinido de $ k $ clusters, minimizando la suma de las varianzas internas de los clusters.
Existen varias implementaciones de este algoritmo, siendo Lloyd's el enfoque más común. En la bibliografía, la varianza interna de los clusters se menciona frecuentemente como "inertia", "within-cluster sum-of-squares" o "varianza intra-cluster".
Consideremos los conjuntos $ C_1, \dots, C_k $, que representan los índices de las observaciones en cada uno de los clusters. Por ejemplo, $ C_1 $ contiene los índices de las observaciones agrupadas en el primer cluster. Se utiliza la notación $ i \in C_k $ para indicar que la observación $ i $ pertenece al cluster $ k $. Estos conjuntos cumplen dos propiedades:
- $ C_1 \cup C_2 \cup \dots \cup C_k = \{1, \dots, n\} $: toda observación está asignada a un cluster.
- $ C_i \cap C_j = \emptyset $ para todo $ i \neq j $: los clusters son disjuntos; una observación no puede pertenecer a más de un cluster a la vez.
El objetivo del algoritmo es minimizar la suma de las distancias cuadradas entre cada observación y el centroide de su cluster. Matemáticamente, esto se expresa como:
$$ (P) \ \text{Minimizar } f(C_l, \mu_l) = \sum_{l=1}^k \sum_{x_n \in C_l} ||x_n - \mu_l ||^2 \text{, respecto a } C_l, \mu_l, $$
donde $ C_l $ es el conjunto de observaciones en el cluster $ l $-ésimo y $ \mu_l $ es su centroide.

Algoritmo¶
- Establecer el número de clusters $ k $.
- Seleccionar aleatoriamente $ k $ observaciones del conjunto de datos como centroides iniciales.
- Asignar cada observación al centroide más cercano.
- Recalcular el centroide de cada cluster.
Repetir los pasos 3 y 4 hasta que las asignaciones no cambien o se alcance el número máximo de iteraciones establecido.
Este problema es NP-hard, lo que significa que es computacionalmente difícil de resolver en tiempo polinomial, colocándolo entre los problemas más complejos de la clase NP.
Ventajas y desventajas¶
K-means es un método de clustering rápido y sencillo, ampliamente utilizado, aunque presenta algunas limitaciones:
Número de clusters: Requiere especificar $ k $ de antemano, lo que puede ser desafiante sin información adicional. Métodos como "elbow" o "silhouette" pueden orientar, pero son aproximados.
Forma de los clusters: Funciona mejor con clusters esféricos, fallando en detectar formas alargadas o irregulares.
Sensibilidad a la inicialización: Los resultados pueden variar según la asignación inicial de centroides; se recomienda repetir el proceso varias veces para obtener mayor estabilidad.
Sensible a outliers: Los valores atípicos pueden afectar significativamente los resultados del clustering.
Aplicación¶
Veamos un ejemplo de análisis no supervisado ocupando el algoritmo k-means.
# librerias
import os
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
from sklearn.datasets import make_blobs
pd.set_option('display.max_columns', 500) # Ver más columnas de los dataframes
# Ver gráficos de matplotlib en jupyter notebook/lab
%matplotlib inline
def init_blobs(N, k, seed=42):
X, y = make_blobs(n_samples=N, centers=k,
random_state=seed, cluster_std=0.60)
return X
# generar datos
data = init_blobs(10000, 6, seed=43)
df = pd.DataFrame(data, columns=["x", "y"])
df.head()
| x | y | |
|---|---|---|
| 0 | -6.953617 | -4.989933 |
| 1 | -2.681117 | 7.583914 |
| 2 | -1.510161 | 4.933676 |
| 3 | -9.748491 | 5.479457 |
| 4 | -7.438017 | -4.597754 |
Al trabajar con distancias, es común que las columnas del dataframe estén en diferentes escalas, lo cual puede afectar el rendimiento de algunos algoritmos, especialmente en sklearn.
Para estos casos, se recomienda normalizar los atributos, es decir, transformar los valores para que estén en una escala acotada o con propiedades específicas. En sklearn existen varias opciones para normalizar:
StandardScaler: Normaliza restando la media y dividiendo por la desviación estándar: $$x_{prep} = \dfrac{x - \mu}{s}$$ Esto garantiza que la media del conjunto sea 0 y la desviación estándar sea 1.
MinMaxScaler: Normaliza utilizando los valores mínimos y máximos del conjunto de datos: $$x_{prep} = \dfrac{x - x_{min}}{x_{max} - x_{min}}$$ Es útil cuando la desviación estándar $ s $ es muy pequeña, lo que lo hace más robusto que StandardScaler en estos casos.
from sklearn.preprocessing import StandardScaler
scaler = StandardScaler()
columns = ['x', 'y']
df[columns] = scaler.fit_transform(df[columns])
df.head()
| x | y | |
|---|---|---|
| 0 | -0.579033 | -1.831435 |
| 1 | 0.408821 | 1.194578 |
| 2 | 0.679560 | 0.556774 |
| 3 | -1.225241 | 0.688121 |
| 4 | -0.691032 | -1.737053 |
# comprobar resultados del estimador
df.describe()
| x | y | |
|---|---|---|
| count | 1.000000e+04 | 1.000000e+04 |
| mean | 2.060574e-16 | -2.285105e-15 |
| std | 1.000050e+00 | 1.000050e+00 |
| min | -1.638247e+00 | -2.410317e+00 |
| 25% | -8.015576e-01 | -4.418042e-01 |
| 50% | -2.089351e-01 | 1.863259e-01 |
| 75% | 5.480066e-01 | 8.159808e-01 |
| max | 2.243358e+00 | 1.639547e+00 |
Con esta parametrización procedemos a graficar nuestros resultados:
# graficar
sns.set(rc={'figure.figsize':(11.7,8.27)})
ax = sns.scatterplot( data=df,x="x", y="y")
Ajustaremos el algoritmo KMeans de sklearn. Los hiperparámetros clave son:
- n_clusters: Número de clusters, o $ K $. El valor predeterminado es 8.
- init: Método de inicialización de centroides.
kmeans++(predeterminado) mejora la calidad inicial, superando a la inicialización aleatoria. - n_init: Número de ejecuciones del algoritmo con diferentes inicializaciones, eligiendo la que minimiza la inercia.
- max_iter: Máximo de iteraciones por ejecución antes de detenerse.
- random_state: Semilla para reproducir los resultados.
- tol: Tolerancia para el criterio de convergencia; valores mayores detienen el algoritmo más rápido.
# ajustar modelo: k-means
from sklearn.cluster import KMeans
X = np.array(df)
kmeans = KMeans(n_clusters=6,n_init=25, random_state=123)
kmeans.fit(X)
centroids = kmeans.cluster_centers_ # centros
clusters = kmeans.labels_ # clusters
# etiquetar los datos con los clusters encontrados
df["cluster"] = clusters
df["cluster"] = df["cluster"].astype('category')
centroids_df = pd.DataFrame(centroids, columns=["x", "y"])
centroids_df["cluster"] = [1,2,3,4,5,6]
# graficar los datos etiquetados con k-means
fig, ax = plt.subplots(figsize=(11, 8.5))
sns.scatterplot( data=df,
x="x",
y="y",
hue="cluster",
legend='full',
palette="Set2")
sns.scatterplot(x="x", y="y",
s=100, color="black", marker="x",
data=centroids_df)
plt.show()
La pregunta natural es: ¿cómo elegir el mejor número de clusters?
No existe un criterio objetivo universal, pero se han desarrollado métodos para orientar esta elección, entre ellos:
- Método del codo (elbow method)
- Criterio de Calinski-Harabasz
- Affinity Propagation (AP)
- Método del gap (y su versión estadística)
- Dendrogramas, entre otros.
Método del Codo¶
Este enfoque evalúa la función de pérdida $ f(C_l, \mu_l) $ al aplicar K-means con diferentes cantidades de clusters, de 1 a $ N $. Al graficar la pérdida en función del número de clusters, se observa un punto de inflexión con forma de "codo". Este punto marca el número óptimo de clusters, donde agregar más clusters aporta cada vez menos reducción en la pérdida.
# implementación de la regla del codo
Nc = range(1, 15)
kmeans = [KMeans(n_clusters=i, n_init=10) for i in Nc] # Suppressing the warning here
score = [kmeans[i].fit(df).inertia_ for i in range(len(kmeans))]
df_Elbow = pd.DataFrame({'Number of Clusters': Nc, 'Score': score})
df_Elbow.head()
| Number of Clusters | Score | |
|---|---|---|
| 0 | 1 | 49176.631100 |
| 1 | 2 | 23045.751688 |
| 2 | 3 | 14457.612924 |
| 3 | 4 | 7042.270693 |
| 4 | 5 | 1665.378832 |
# graficar los datos etiquetados con k-means
fig, ax = plt.subplots(figsize=(11, 8.5))
plt.title('Elbow Curve')
sns.lineplot(x="Number of Clusters",
y="Score",
data=df_Elbow)
sns.scatterplot(x="Number of Clusters",
y="Score",
data=df_Elbow)
plt.show()
A partir de 4 clusters la reducción en la suma total de cuadrados internos parece estabilizarse, indicando que $k$ = 4 es una buena opción.
Hierarchical clustering¶
El Hierarchical Clustering es un método de agrupamiento no supervisado que organiza los datos en una estructura jerárquica representada por un dendrograma. Existen dos enfoques principales:
- Aglomerativo (ascendente): parte de cada observación como un cluster y los fusiona progresivamente hasta formar uno solo.
- Divisivo (descendente): comienza con un único cluster que contiene todas las observaciones y lo divide hasta llegar a clusters individuales.
El algoritmo depende de:
Métrica de distancia: mide la similitud entre observaciones (euclidiana, Manhattan, etc.).
Función de enlace: define cómo calcular la distancia entre clusters al fusionarlos o dividirlos:
- Completo: distancia máxima entre puntos.
- Simple: distancia mínima.
- Promedio: distancia media.
- Ward: minimiza la varianza dentro de los clusters.
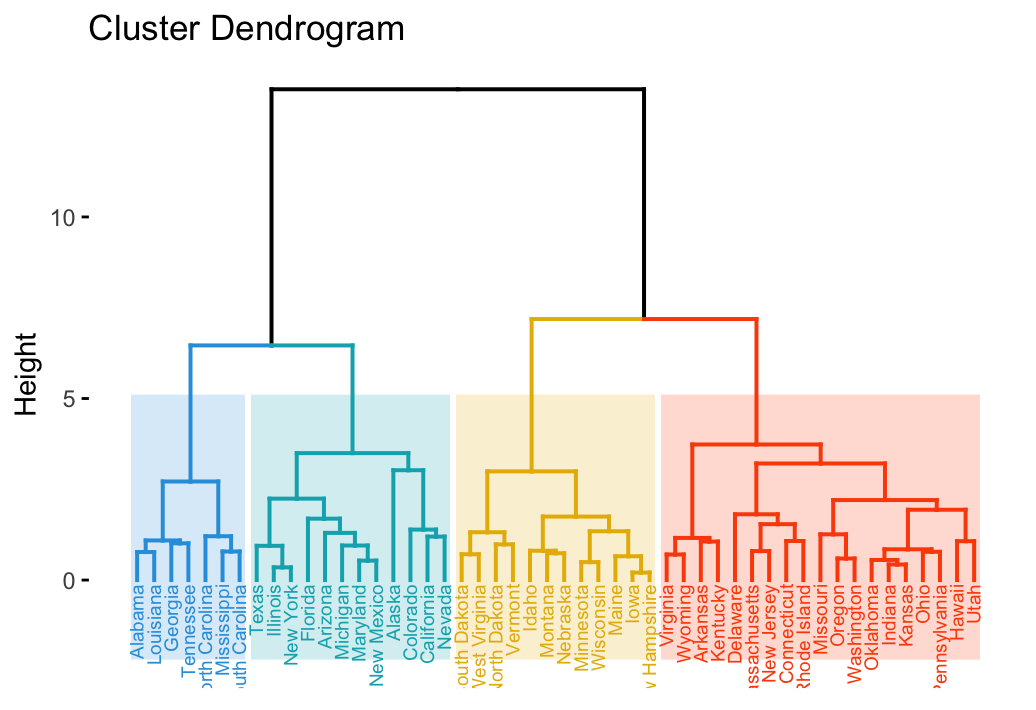
Dendrograma
El dendrograma es la representación gráfica de la estructura generada por el hierarchical clustering. Se muestra como un árbol donde se visualiza el proceso de fusión o división de los clusters a lo largo del algoritmo.
Interpretación
- Cada hoja del dendrograma corresponde a una observación.
- Cada rama indica la unión de dos clusters o puntos individuales.
- La altura de la unión refleja la distancia o disimilitud entre los clusters, determinada por la métrica de distancia y la función de enlace utilizadas.
En términos matemáticos, esta altura representa el nivel de similitud: cuanto más baja es la unión, más parecidos son los clusters.
Selección del número de clusters
Para decidir cuántos clusters obtener, se realiza un corte horizontal en el dendrograma a una cierta altura. Cada grupo de ramas separado por ese corte constituye un cluster. Generalmente, se elige el punto donde hay un salto notable en la altura entre fusiones consecutivas, lo que indica un cambio importante en la disimilitud entre los datos.
Aplicación¶
En este ejemplo, generaremos un conjunto de datos sintético y aplicaremos Hierarchical Clustering para analizar su estructura. Nuestro objetivo es agrupar las observaciones en clusters y visualizar el proceso jerárquico a través de un dendrograma, identificando el número óptimo de clusters mediante un punto de corte.
import matplotlib.pyplot as plt
from sklearn.datasets import make_blobs
from scipy.cluster.hierarchy import dendrogram, linkage
import numpy as np
# Generar datos
X, y = make_blobs(
n_samples=200,
n_features=2,
centers=4,
cluster_std=0.60,
shuffle=True,
random_state=0
)
# Aplicar el clustering jerárquico y calcular los enlaces
Z = linkage(X, method='ward') # 'ward' minimiza la varianza dentro de los clusters
# Determinar el punto de corte (por ejemplo, a una altura de 6)
cut_height = 6
# Graficar el dendrograma con una línea de corte
plt.figure(figsize=(10, 7))
dendrogram(Z)
plt.axhline(y=cut_height, color='red', linestyle='--', label=f'Corte a altura = {cut_height}')
plt.title("Dendrograma del Clustering Jerárquico")
plt.xlabel("Observaciones")
plt.ylabel("Distancia")
plt.legend()
plt.show()
Density-Based Clustering (DBSCAN)¶
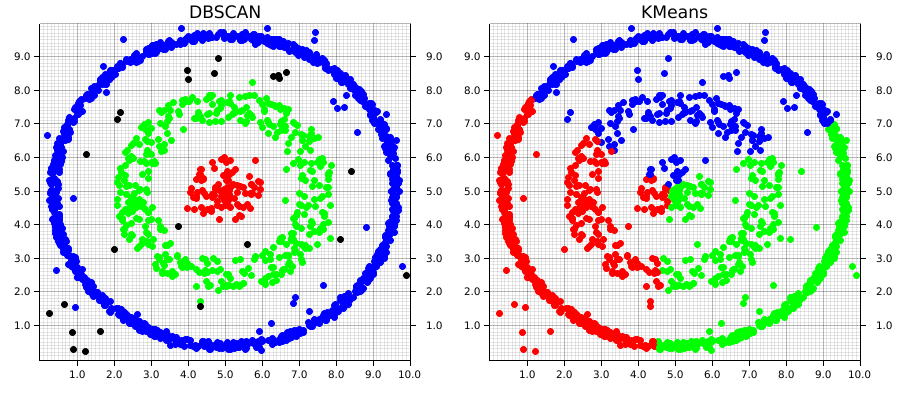
El DBSCAN (Density-Based Spatial Clustering of Applications with Noise) es un algoritmo de clustering basado en densidad que agrupa puntos según su proximidad local. A diferencia de métodos como K-means, no requiere fijar de antemano el número de clusters, puede detectar formas arbitrarias y manejar ruido u outliers.
Conceptos clave
- Radio de vecindad ($\varepsilon$): distancia máxima para considerar a otro punto como vecino.
- Mínimo de puntos (MinPts): número mínimo de vecinos para que una región sea considerada densa y forme un cluster.
Funcionamiento
Clasificación de puntos:
- Centrales: tienen al menos MinPts en su vecindad.
- De borde: están cerca de un punto central, pero no cumplen la condición por sí solos.
- Ruido: no pertenecen a ningún cluster.
Expansión de clusters: a partir de un punto central se agregan vecinos y se repite el proceso recursivamente.
Identificación de ruido: los puntos que no pueden asignarse a ningún cluster quedan como anomalías.
Ventajas y desventajas
- Ventajas: no necesita definir $k$, detecta clusters no esféricos y encuentra outliers.
- Desventajas: depende fuertemente de los parámetros $\varepsilon$ y MinPts, y puede fallar en datos con densidades muy variables.
DBSCAN se aplica en contextos como detección de anomalías y análisis geoespacial, donde los clusters no siguen formas regulares.
Aplicación¶
Para este ejemplo, generaremos un conjunto de datos sintético con grupos de puntos en diferentes regiones, usando la función make_moons de sklearn.datasets. Este conjunto de datos produce dos clusters con forma de media luna, que son difíciles de separar mediante métodos de clustering tradicionales como K-means, ya que los clusters no son esféricos y están entrelazados.
# Importar bibliotecas necesarias
import pandas as pd
import matplotlib.pyplot as plt
from sklearn.datasets import make_moons
from sklearn.cluster import DBSCAN
# Generar el conjunto de datos
n_muestras = 300
ruido = 0.1
semilla = 42
X, y = make_moons(n_samples=n_muestras, noise=ruido, random_state=semilla)
# Crear un DataFrame para los datos
df = pd.DataFrame(X, columns=['Feature1', 'Feature2'])
# Ajustar DBSCAN con un eps mayor y menos min_samples
eps = 0.18
min_muestras = 4
dbscan = DBSCAN(eps=eps, min_samples=min_muestras)
# Aplicar DBSCAN a los datos
df['Cluster'] = dbscan.fit_predict(df[['Feature1', 'Feature2']])
# Visualización de resultados
plt.figure(figsize=(10, 6))
colores = ['blue', 'green', 'yellow', 'purple', 'orange']
for cluster in df['Cluster'].unique():
if cluster == -1:
color, label = 'red', 'Ruido (Outliers)'
else:
color, label = colores[cluster % len(colores)], f'Cluster {cluster + 1}'
plt.scatter(df[df['Cluster'] == cluster]['Feature1'],
df[df['Cluster'] == cluster]['Feature2'],
s=50, color=color, label=label)
plt.title(f"Clustering Basado en Densidad (DBSCAN) - Ajuste de Parámetros (eps={eps}, min_samples={min_muestras})")
plt.xlabel("Característica 1")
plt.ylabel("Característica 2")
plt.legend()
plt.show()
Gaussian Mixture Models (GMMs)¶
Los Gaussian Mixture Models (GMMs) son un método de clustering probabilístico que asume que los datos provienen de una combinación de varias distribuciones gaussianas. Cada cluster se modela con su propia media, varianza y peso, permitiendo que los clusters se solapen, a diferencia de métodos como K-means.
Conceptos clave
- Distribución mixta: los datos se generan a partir de $K$ gaussianas.
- Asignación probabilística: cada punto tiene una probabilidad de pertenecer a cada cluster, en lugar de una asignación exclusiva.
- Parámetros: media ($\mu$), varianza ($\sigma^2$) y peso del cluster ($\pi$).
Algoritmo EM (Expectation-Maximization)
- Paso E: calcula la probabilidad de que cada punto pertenezca a cada gaussiana.
- Paso M: actualiza los parámetros para maximizar la verosimilitud de los datos. El proceso se repite hasta la convergencia.
Aplicaciones: Los GMMs son útiles cuando los datos presentan clusters elípticos o solapados, y se aplican en segmentación de datos, detección de anomalías y reducción de dimensionalidad.
Aplicación¶
En este ejemplo, generaremos un conjunto de datos sintético utilizando la función make_blobs de sklearn. Este conjunto de datos consistirá en 300 puntos distribuidos en torno a tres centros o clusters, con una dispersión específica. Utilizaremos GMM para identificar los clusters, lo cual es especialmente útil cuando los clusters tienen forma elíptica o se superponen ligeramente.
import pandas as pd
import matplotlib.pyplot as plt
from sklearn.datasets import make_blobs
from sklearn.mixture import GaussianMixture
# Generar el conjunto de datos
X, y = make_blobs(n_samples=300, centers=3, cluster_std=1.0, random_state=0)
# Crear un DataFrame para los datos
df = pd.DataFrame(X, columns=['Feature1', 'Feature2'])
# Aplicar Gaussian Mixture Model
gmm = GaussianMixture(n_components=3, random_state=0)
df['Cluster'] = gmm.fit_predict(df[['Feature1', 'Feature2']])
# Visualización de resultados
plt.figure(figsize=(10, 6))
for cluster in range(gmm.n_components):
plt.scatter(df[df['Cluster'] == cluster]['Feature1'],
df[df['Cluster'] == cluster]['Feature2'],
s=50, label=f'Cluster {cluster + 1}')
# Graficar las medias de cada componente (centroides)
plt.scatter(gmm.means_[:, 0], gmm.means_[:, 1], s=200, c='black', marker='X', label='Centroides')
plt.title("Clustering con Gaussian Mixture Models (GMM)")
plt.xlabel("Característica 1")
plt.ylabel("Característica 2")
plt.legend()
plt.show()